基于昇思(MindSpore)的深度学习疟疾检测——疟疾病理切片的判读(有手就行的新手入门)
本项目的完成我必须十分感谢我的teammate @lisenlingood,非常感谢带我入门深度学习以及各种开发知识。
项目主要面向新手小白的入门级小项目,本项目从原理开始,附带上了详细的系统环境的搭建过程以及源代码(环境配置永远的噩梦/(ㄒoㄒ)/~~)。
主要使用的框架为基于昇思(MindSpore)的Vision Transformer(VIT)。
本项目完全开源,使用MIT开源协议。
在下载资源后可以先阅读readme,里面带有文件的使用说明
网盘网址附在文末。
吐槽:期末考试来了,学不完,根本学不完 ,怎么想都学不完吧w(゚Д゚)w,要疯了。。。
一、项目设计引言
1.1研究背景
迄今疟疾在全球范围内的流行仍很严重,世界人口约有40%生活在疟疾流行区域。疟疾仍是非洲大陆上最严重的疾病,约有5亿人口生活在疟疾流行区,每年全球约有1亿人有疟疾临床症状,其中90%的患者在非洲大陆,每年死于疟疾的人数超过200万。亚洲东南部,中部也是疟疾流行猖獗的地区。中南美洲仍有疟疾流行。
对于非洲大部分地区仍处于贫穷的状态,卫生条件的不足和医疗设备的落后导致造成对疟疾等相关疾病的识别能力不足,常不能及时检测或不能准确检测。随着人工智能的快速发展,计算机辅助医学图像分析技术正在不断发展且完善。在实际的临床使用中能够有效解决欠发达地区医疗水平不足的弊端。通过引入机器学习和深度学习方法,用计算机辅助判断疟疾正在逐步成为现实。
1.2研究目的
通过使用疟疾患者的薄血膜图片作为数据集进行深度学习训练,可以得到判断疟疾是否存在的一个训练模型。在实际使用时能够针对待测样本进行图像分析,判断该样本是否属于疟疾病人的样本,可以实现对疟疾的快速诊断。 在欠发达地区,这项技术可为当地群众提供准确快速的疟疾诊断,弥补当地医疗资源短缺和人手不足的问题。能有效进行疟疾的诊断同时也为后续的治疗提供帮助。
二、研究设计过程
2.1模型原理
2.1.1简介
在本项目中使用的模型为Vision Transformer(VIT),以下简称为VIT。ViT是2020年Google团队提出的将Transformer应用在图像分类的模型,虽然不是第一篇将transformer应用在视觉任务的论文,但是因为其模型"简单"且效果好,可扩展性强(scalable,模型越大效果越好),成为了transformer在CV领域应用的里程碑著作。ViT是自然语言处理和计算机视觉两个领域的融合结晶。在不依赖卷积操作的情况下,依然可以在图像分类任务上达到很好的效果。
2.1.2模型结构
ViT模型的主体结构是基于Transformer模型的Encoder部分(部分结构顺序有调整,如:Normalization的位置与标准Transformer不同),其结构图如下:
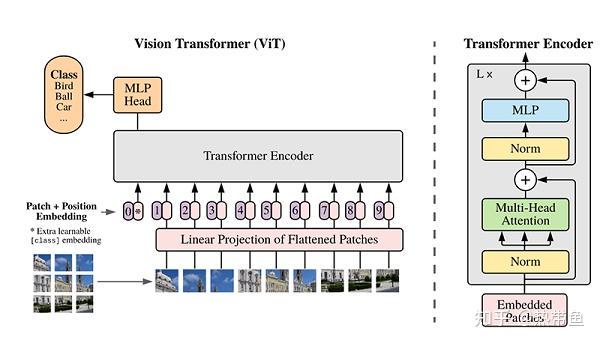

2.1.3模型特点
ViT模型主要应用于图像分类领域。因此,其模型结构相较于传统的Transformer有以下几个特点: 数据集的原图像被划分为多个patch后,将二维patch(不考虑channel)转换为一维向量,再加上类别向量与位置向量作为模型输入。 模型主体的Block结构是基于Transformer的Encoder结构,但是调整了Normalization的位置,其中,最主要的结构依然是Multi-head Attention结构。 模型在Blocks堆叠后接全连接层,接受类别向量的输出作为输入并用于分类。通常情况下,我们将最后的全连接层称为Head,Transformer Encoder部分为backbone。
2.1.4模型解析
Transformer模型源于2017年的一篇文章。在这篇文章中提出的基于Attention机制的编码器-解码器型结构在自然语言处理领域获得了巨大的成功。模型结构如下图所示:


其主要结构为多个Encoder和Decoder模块所组成,其中Encoder和Decoder的详细结构如下图所示: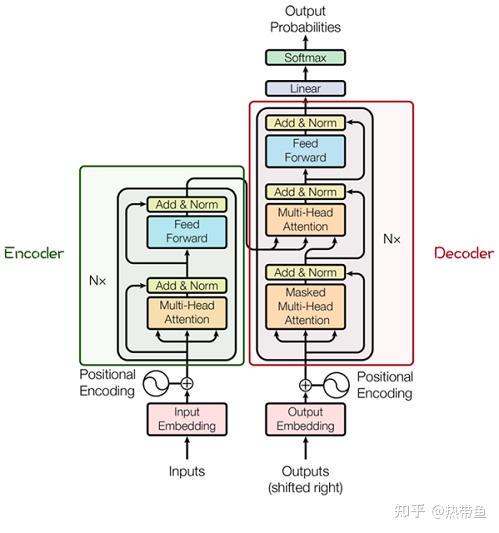

Encoder与Decoder由许多结构组成,如:多头注意力(Multi-Head Attention)层,Feed Forward层,Normaliztion层,甚至残差连接(Residual Connection,图中的"Add")。不过,其中最重要的结构是多头注意力(Multi-Head Attention)结构,该结构基于自注意力(Self-Attention)机制,是多个Self-Attention的并行组成。
2.2模型训练及效果
2.2.1模型训练
该模型训练所用数据集来自于National Library of Medicine,以下为数据集下载链接https://lhncbc.nlm.nih.gov/LHC-research/LHC-projects/image-processing/malaria-datasheet.html。获得数据集后需根据mindspore的数据集进行预处理。在导入数据后,需要构建VIT模型。以下为模型构建流程示意图:
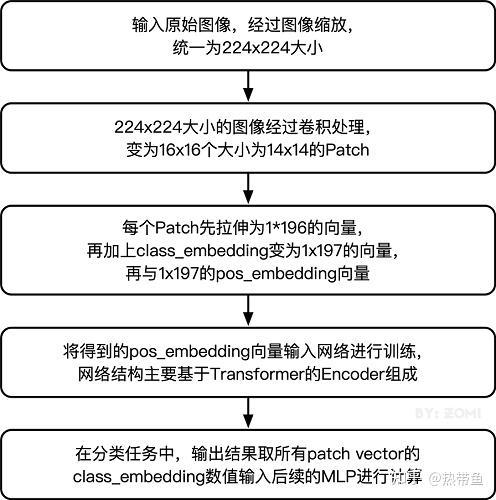

完整训练ViT模型需要很长的时间,实际应用时建议根据项目需要调整epoch_size。本次项目中受限于算力,相关参数的设定epoch_size = 10、momentum = 0.9、num_classes = 1000。以下为训练过程展示:

2.2.2模型效果
1、该模型使用业界通用的评价标准Top_1_Accuracy和Top_5_Accuracy评价指标来评价模型表现。该评价标准定义为: Top-1: Accuracy是指排名第一的类别与实际结果相符的准确率,就是你预测的label取最后概率向量里面最大的那一个作为预测结果,如过你的预测结果中概率最大的那个分类正确,则预测正确。否则预测错误。 Top-5: Accuracy是指排名前五的类别包含实际结果的准确率,就是最后概率向量最大的前五名中,只要出现了正确概率即为预测正确。否则预测错误。
以下为本项目的模型评价结果
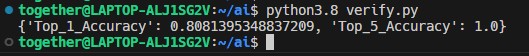

通过上图可知:Top_1_Accuracy=0.8081、Top_5_Accuracy=1.0。可知该模型具有优良的准确性。
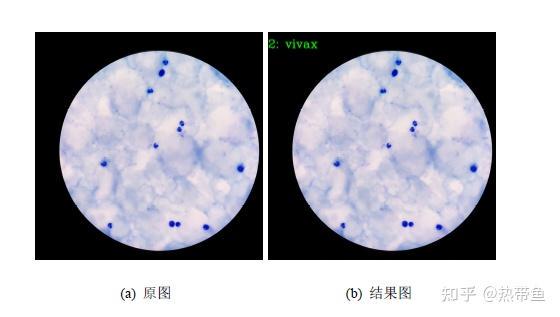
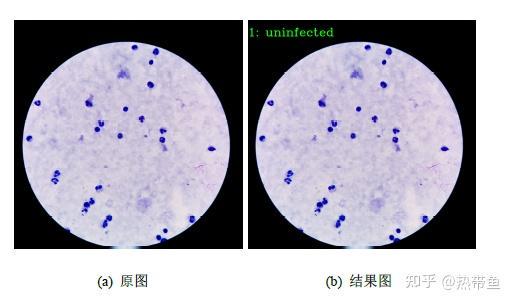
2、输出效果图展示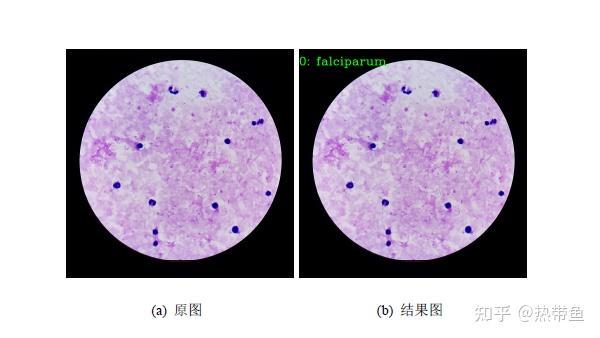



三、项目环境搭建
3.1wsl下Ubuntu的安装与基础环境配置
3.1.1启用适用于linux的windows子系统
以管理员身份打开 PowerShell("开始"菜单 >"PowerShell" >单击右键 >"以管理员身份运行"),然后输入以下命令:

3.1.2检查运行 WSL 2 的要求
对于 x64 系统:版本 1903 或更高版本,内部版本为 18362.1049或更高版本 对于 ARM64 系统:版本 2004 或更高版本,内部版本为 19041 或更高版本 若要更新到 WSL 2,需要 Windows 10或Windows 11
3.1.3启用虚拟机功能
安装 WSL 2 之前,必须启用"虚拟机平台"可选功能。 计算机需要虚拟化功能才能使用此功能。以管理员身份打开 PowerShell 并运行:
3.1.4将 WSL 2 设置为默认版本
打开 PowerShell,然后在安装新的 Linux 发行版时运行以下命令,将 WSL 2 设置为默认版本:
3.1.5安装所选的 Linux 分发
打开 Microsoft Store,并选择你偏好的 Linux 分发版:本次使用的是Ubuntu 20.04.6 LST。


3.1.6创建用户帐户和密码
首次启动新安装的 Linux 分发版时,将打开一个控制台窗口,系统会要求你等待一分钟或两分钟,以便文件解压缩并存储到电脑上。 未来的所有启动时间应不到一秒。然后,需要为新的 Linux 分发版创建用户帐户和密码。

3.1.7Vscode配置
为了更加方便的使用wsl,本次项目中使用了vscode作为WSL的编辑器,首先需安装vscode并在拓展中下载WSL拓展。

以下为运行页面展示:


3.1.8创建用户帐户和密码
首次进入Ubuntu使先需获取root权限: 在终端中输入
输入所要设置的密码即可获取root权限。
3.1.9安装pip
想要在 Ubuntu 20.04 上为 Python 3 安装 pip,以 root 或者其他 sudo 用户身份在终端运行下面的命令:
上面的命令将会安装用来构建 Python 模块所需要的所有依赖软件包。 当安装结束,验证安装过程,检查 pip 版本:
以下为效果展示:

3.1.10安装python3.8
安装依赖包
添加 deadsnakes PPA 源
删除原来的软连接
添加新的软连接,可以用which python3.8来看一下python3.8可执行文件在哪个位置
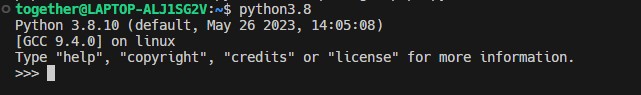
在终端中输入python3.8,此时显示你的python3.8的版本

3.2mindspore以及相关环境配置
3.2.1检查显卡驱动
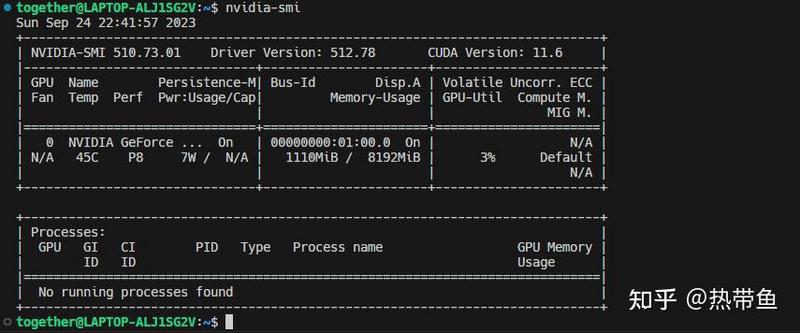
CUDA 10.1要求最低显卡驱动版本为418.39;CUDA 11.1要求最低显卡驱动版本为450.80.02;CUDA 11.6要求最低显卡驱动为510.39.01。执行以下指令检查驱动版本:
以下为输出展示:

3.2.2gcc安装
默认的 Ubuntu 软件源包含了一个软件包组,名称为 "build-essential",它包含了 GNU 编辑器集合,GNU 调试器,和其他编译软件所必需的开发库和工具。本次项目安装的版本为:gcc (Ubuntu 9.4.0-1ubuntu1 20.04.2) 9.4.0
想要安装开发工具软件包,以 拥有 sudo 权限用户身份或者 root 身份运行下面的命令:
这个命令将会安装一系列软件包,包括gcc,g++,和make。你可能还想安装关于如何使用 GNU/Linux开发的手册。
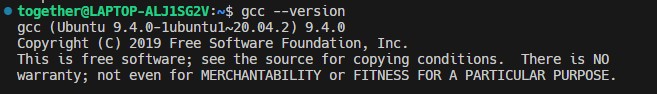
通过运行下面的命令,打印 GCC 版本,来验证 GCC 编译器是否被成功地安装。
以下为输出展示:

3.2.3CUDA安装
在安装CUDA前需要先安装相关依赖,执行以下命令:
CUDA 10.1要求最低显卡驱动版本为418.39;CUDA 11.1要求最低显卡驱动版本为450.80.02;CUDA 11.6要求最小的显卡驱动版本为510.39.01。可以执行nvidia-smi指令确认显卡驱动版本。如果驱动版本不满足要求,CUDA安装过程中可以选择同时安装驱动,安装驱动后需要重启系统。使用以下命令安装CUDA 11.6(推荐)。
更新一下可能需要的依赖
用以下代码检查cuda是否检查成功,注意此处的cuda版本是你在toolkit下载那里决定的,这个版本可能和nvidia-smi显示的版本号不一样,可高可低,这是因为nvidia给cuda开了两个api,这两个api只要差不是太远,基本都可以保证正常运行cuda
以下为输出展示:

3.2.4cuDNN安装
完成CUDA的安装后,在cuDNN页面登录并下载对应的cuDNN安装包。如果之前安装了CUDA 10.1,下载配套CUDA 10.1的cuDNN v7.6.x;如果之前安装了CUDA 11.1,下载配套CUDA 11.1的cuDNN v8.0.x;如果之前安装了CUDA 11.6,下载配套CUDA 11.6的cuDNN v8.5.x。注意下载后缀名为tgz的压缩包。假设下载的cuDNN包名为cudnn.tgz,安装的CUDA版本为11.6,执行以下命令安装cuDNN:
如果之前安装了其他CUDA版本或者CUDA安装路径不同,只需替换上述命令中的/usr/local/cuda-11.6为当前安装的CUDA路径。
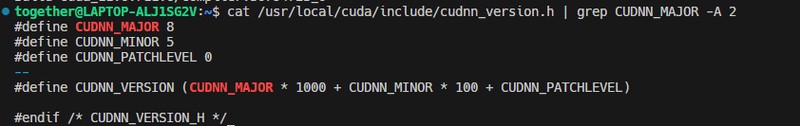
查看cuDNN版本方法:
以下为输出展示:

3.2.5mindspore安装
首先在mndspore官网安装页面http://mindspore.cn/install/寻找合适计算机系统的版本。
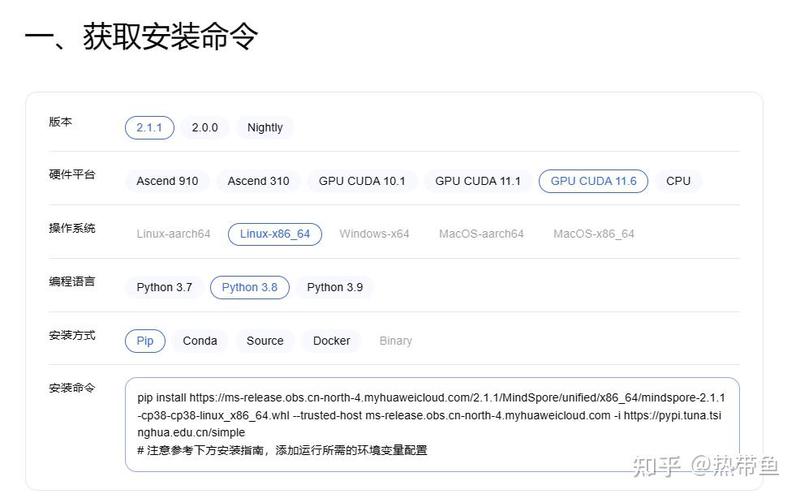

注意,本次项目使用的版本为2.1.0,以下为pip安装命令:
验证是否成功安装 运行MindSpore GPU版本前,请确保nvcc的安装路径已经添加到环境变量中,如果没有添加,以安装在默认路径的CUDA11为例,可以执行如下操作:
若输出以下结果则说明Mindspore已经成功安装。


四、项目使用方法
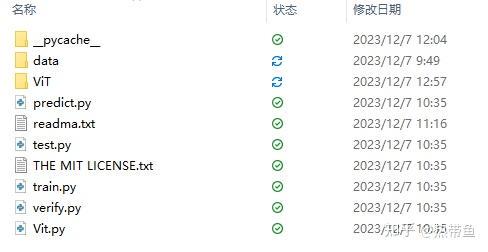

1、本项目结构如文件所示。
data文件夹即数据集,里面包含测试集test以及数据集train
2、VIT文件夹即网页中展示的已经训练好的模型
3、python文件:如文件名字所示,test即为测试程序,train即为训练程序,verify即为验证程序。
由于时间太长没有碰了,predict和vit有点忘了,不过估计是预处理或者模型处理的程序吧,各
位可以跑跑看有什么效果。
4、如果各位是新手,记得在使用前修改其中的文件地址,一般情况下,报错大概率是由于环境配置
错误(版本错误)或者文件地址修改错误(漏了地址没有修改或者/ \两个符号没有变换)等等。
5、如果有问题欢迎交流
6、本项目完全开源,使用了MIT协议即:允许他人修改源代码后再闭源,不用对修改过的文件做说明,
且二次开发的软件可以使用原作者的名字做营销。
The MIT license:
Copyright <2023> <lisenlingood、热带鱼>
Permission is hereby granted, free of charge, to any person obtaining a copy of this software
and associated documentation files (the “Software”), to deal in the Software without restric
tion, including without limitation the rights to use, copy, modify, merge, publish, distribute, sub
license, and/or sell copies of the Software, and to permit persons to whom the Software is furni
shed to do so, subject to the following conditions:
The above copyright notice and this permission notice shall be included in all copies or substantia
l portions of the Software.
THE SOFTWARE IS PROVIDED “AS IS”, WITHOUT WARRANTY OF ANY KIND, EXPRESS OR IMPLIED
, INCLUDING BUT NOT LIMITED TO THE WARRANTIES OF MERCHANTABILITY, FITNESS FOR A PART
ICULAR PURPOSE AND NONINFRINGEMENT. IN NO EVENT SHALL THE AUTHORS OR COPYRIGHT H
OLDERS BE LIABLE FOR ANY CLAIM, DAMAGES OR OTHER LIABILITY, WHETHER IN AN ACTION OF C
ONTRACT, TORT OR OTHERWISE, ARISING FROM, OUT OF OR IN CONNECTION WITH THE SOFTWA
RE OR THE USE OR OTHER DEALINGS IN THE SOFTWARE.
五、总结
通过华为昇思平台mindspore实现本项目的数据处理、模型训练、模型推理等等内容。通过本次项目所训练出的模型评价为Top_1_Accuracy=0.8081、Top_5_Accuracy=1.0。可知该模型具有优良的准确率。当输入图片进行推理时可以看出其预测的结果。
通过本次基于深度学习的疟疾病理切片的判读项目的学习与时间,掌握了Ubuntu系统的使用、深度学习计算机环境的搭建以及学习了vision Transformer框架。通过华为昇思平台mindspore实现本项目的数据处理、模型训练、模型推理等等内容。通过本次项目所训练出的模型评价为Top_1_Accuracy=0.8081、Top_5_Accuracy=1.0。

六、展望
该项目可以根据昇思(mindespore)搭载于硬件开发板上进行推理应用,具体可以根据官网文档进行操作,这里由于时间关系没有制作,如果有感兴趣的读者可以进行深度的开发。
资料
百度网盘:
1、文件夹版本:链接:https://pan.baidu.com/s/1KIUFvrtwHY1bfcW-v23c7Q?pwd=2gf8
提取码:2gf8
2、压缩包版本:链接:https://pan.baidu.com/s/1fKPlT2OQfNcImE7o3AaFGQ?pwd=zryc
提取码:zryc
参考:1、https://www.mindspore.cn/tutorials/application/zh-CN/r2.1/cv/vit.html
2、MindSpore
3、MindSpore官网
 Merry Christmas
Merry Christmas
个人能力有限,如有纰漏,欢迎指正。
如有内容或图片侵权,请联系删除。
